Análisis de metilación en pacientes con leucemia mieloide crónica en diferentes instituciones médicas de Medellín
Resumen
Fundamento: la leucemia mieloide crónica es una neoplasia mieloproliferativa crónica que se caracteriza por la presencia del cromosoma filadelfia. Para esta se ha diseñado como tratamiento los inhibidores de tirosin kinasa, que genera en los pacientes una respuesta hematológica, citogenética y molecular. Esta enfermedad se caracteriza por presentar tres fases clínicas que son producto de la acumulación de daños tanto genéticos representados por mutaciones puntuales y alteraciones en el cariotipo; y cambios epigenéticos en genes tales como ABL-1, OSCP1, PDLIM4, NPM2, ER y p15.
Objetivo: describir los patrones de metilación de seis genes en pacientes con leucemia mieloide crónica en distintas fases de la enfermedad tratados con algún ITK´s o en su defecto con hidroxiurea en dos hospitales de Medellín.
Métodos: se realizó un estudio analítico trasversal, en el que se recolectaron 34 muestras a conveniencia de pacientes con leucemia mieloide crónica. Para hacer el análisis de estas muestras se usó una PCR específica de metilación. Los datos analizados no se distribuyeron de forma normal, por lo que se realizaron pruebas no paramétricas como U de Man Whitney y test exacto de Fischer, con una significancia de 0,05.
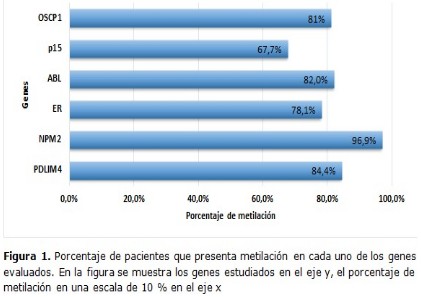
Resultados: se encontró diferencias de estadísticas significativas en los datos del hemograma de ambas fases de la enfermedad, también se encontró una alta frecuencia de genes metilados en fase acelerada. La metilación de p15, ABL-1, ER son independiente de la fase de la enfermedad. En los pacientes tratados con hidroxiurea se observó una metilación del 100 % para los genes NPM2, OSCP1 y PDLIM4 este comportamiento no se observó en los individuos tratados con ITK´S, no obstante, para aquellos pacientes que desarrollaron resistencia a los ITK´s se observó un porcentaje de metilación mayor en los genes OSCP1, ABL-1 y PDLIM4.
Conclusiones: la metilación en los genes PDLIM4 y OSCP1, puede asociarse con pronóstico desfavorable, ya que puede estar relacionado con la progresión de fase crónica a acelerada y el desarrollo de resistencia a los ITK´s.
DeCS: LEUCEMIA MIELÓGENA CRÓNICA BCR-ABL POSITIVA; METILACIÓN; CROMOSOMA FILADELFIA; HIDROXIUREA/uso terapéutico; ESTUDIOS TRANSVERSALES.
Palabras clave
Referencias
JW_Vardiman JV, Baccarani J MM. Thiele. Chronic myelogenous leukaemia, BCR-ABL1 positive In: Jaffe ES, Harris NL, Stein H, Vardiman JW, editors.World Health Organization (WHO) Classification of Tumours. Pathology & Genetics. Tumours of Haematopoietic and Lymphoid Tissues. Lyon, France: IARC Press; 2001. p. 291-302.
Howlader N, Noone AM, Krapcho M, Miller D, Bishop K, Altekruse SF, et al, editors. SEER Cancer Statistics Review, 1975-2013 [Internet]. Bethesda: National Cancer Institute; 2016 Apr [cited 2016 May 12]. Available from: http://seer.cancer.gov/csr/1975_2013/.
Ferlay J, Soerjomataram I, Ervik M, Dikshit R, Eser S, Mathers C, et al. Cancer Incidence and Mortality. GLOBOCAN [Internet]. 2012 [cited 2016 May 12]1(1):[about 8 p.]. Available from: http://globocan.iarc.fr/old/summary_table_sitehtml.asp?selection=12280&title=Leukaemia&sex=0&type=0&window=1&america=2&sort=3&submit=%C2%A0Execute accessed on 23/05/2016.
Höglund M, Sandin F, Simonsson B. Epidemiology of chronic myeloid leukaemia: an update. Ann Hematol. 2015;94(S2):241–7.
National cancer Institute. Chronic Myeloid Leukemia - SEER Stat Fact Sheets [Internet]. SEER Stat Fact Sheets: chronic Myeloid Leukemia (CML); 2016 [citado 2016 May 17]. Available from: http://seer.cancer.gov/statfacts/html/cmyl.html
Direccion de medicamentos y tecnologias en salud. Actuación admistrativa de la declaratoria de razones de interes público. Medellin: Universidad de Antioquia; 2016.
Cilloni D, Saglio G. Molecular pathways: BCR-ABL. Clin Cancer Res. 2012;18(4):930–7.
Huu NT, Yoshida H, Yamaguchi M. Overexpression of tumor suppressor protein OSCP1/NOR1 induces ER stress and apoptosis during development of Drosophila melanogaster. Am J Cancer Res [Internet]. 2015 Jan [2016 citado Feb 29];5(5):[about 11 p.]. Available from: http pmc/articles/PMC4497438/?report=abstract
Asimakopoulos FA, Shteper PJ, Krichevsky S, Fibach E, Polliack A, Rachmilewitz E, et al. ABL1 methylation is a distinct molecular event associated with clonal evolution of chronic myeloid leukemia. Blood. 1999 Oct 1;94(7):2452-60.
Issa J-PJ, Zehnbauer BA, Civin CI, Collector MI, Sharkis SJ, Davidson NE, et al. The Estrogen Receptor CpG Island Is Methylated in Most Hematopoietic Neoplasms. Cancer Res. 1996 Mar 1;56(5):973-7.
Hantschel O, Grebien F, Superti-Furga G. The growing arsenal of ATP-competitive and allosteric inhibitors of BCR-ABL. Cancer Res. 2012;72(19):4890-5.
Comert M, Baran Y, Saydam G. Changes in molecular biology of chronic myeloid leukemia in tyrosine kinase inhibitor era. Am J Blood Res. 2013 Jan;3(3):191-200.
Weisberg E, Manley PW, Cowan-Jacob SW, Hochhaus A, Griffin JD. Second generation inhibitors of BCR-ABL for the treatment of resistant chronic myeloid leukaemia. Nat Rev Cancer [Internet]. 2007 May [citado 2013 Nov 13];7(5):[about 12 p.]. Available from: http://www.ncbi.nlm.nih.gov/pubmed/17457302
Kimura S. [State-of-the-art management of CML in 2015 and future prospects]. Rinsho Ketsueki [Internet]. 2015 Oct [citado 2016 Nov 19];56(10):[about 9 p.]. Available from: http://www.ncbi.nlm.nih.gov/pubmed/26458439
Chen S, Sutiman N. Pharmacogenetics of drug transporters in modulating disposition and treatment outcomes in chronic myeloid leukemia & gastrointestinal stromal tumor patients. Pharmacogenomics. 2016;17(17):1941-55.
Dietrich D, Lesche R, Tetzner R, Krispin M, Dietrich J, Haedicke W, et al. Analysis of DNA Methylation of Multiple Genes in Microdissected Cells From Formalin-fixed and Paraffin-embedded Tissues. J Histochem Cytochem. 2009;57(5):477-89.
Baccarani M, Castagnetti F, Gugliotta G, Rosti G. A review of the European LeukemiaNet recommendations for the management of CML. Ann Hematol [Internet]. abril de 2015 [citado 2016 Apri 1];94 Suppl 2:[about 7 p.]. Available from: http://www.ncbi.nlm.nih.gov/pubmed/25814080
Furtado VF, Santos GR, de Carvalho DS, Staziaki PV, Pasquini R, Funke VAM. Accelerated phase chronic myeloid leukemia: evaluation of clinical criteria as predictors of survival, major cytogenetic response and progression to blast phase. Rev Bras Hematol Hemoter. 2015 Ene;37(5):341-7.
Jelinek J, Gharibyan V, Estecio MRH, Kondo K, He R, Chung W, et al. Aberrant DNA methylation is associated with disease progression, resistance to imatinib and shortened survival in chronic myelogenous leukemia. PLoS One. 2011;6(7):e22110.
Bodoor K, Haddad Y, Alkhateeb A, Al-Abbadi A, Dowairi M, Magableh A, et al. DNA hypermethylation of cell cycle (p15 and p16) and apoptotic (p14, p53, DAPK and TMS1) genes in peripheral blood of leukemia patients. Asian Pacific J Cancer Prev. 2014;15(1):75-84.
Gómez Gómez M, Danglot Banck C, Vega Franco L. Sinopsis de pruebas estadísticas no paramétricas. Cúando usarlas. Rev Mex Pedriatría. 2003;70(2):91-9.
Preisler HD, Li B, Chen H, Fisher L, Nayini J, Raza A, et al. P15 INK4B gene methylation and expression in normal , myelodysplastic , and acute myelogenous leukemia cells and in the marrow cells of cured lymphoma patients. 2001;1589-95.
McCabe MT, Brandes JC, Vertino PM. Cancer DNA methylation: molecular mechanisms and clinical implications. Clin Cancer Res. 2009 Jun 15;15(12):3927-37.
Jiang D, Hong Q, Shen Y, Xu Y, Zhu H, Li Y, et al. The diagnostic value of DNA methylation in leukemia: a systematic review and meta-analysis. PLoS One. 2014 Jan;9(5):e96822.
Berrio Carvajal D, Acevedo Toro PA. Frecuencia de la mutación T315I y su relación con la resistencia a inhibidores tirosina kinasa en pacientes con leucemia mieloide crónica, en Medellín. 2015. Medellin: Universidad de Antioquia; 2016.
Elias MH, Baba AA, Husin A, Sulong S, Hassan R, Sim GA, et al. HOXA4 gene promoter hypermethylation as an epigenetic mechanism mediating resistance to imatinib mesylate in chronic myeloid leukemia patients. Biomed Res Int. 2013 Jan;2013:129715.
Yang H, Liang H, Yan J, Tao R, Hao S, Ma L. Down-regulation of hematopoiesis master regulator PU.1 via aberrant methylation in chronic myeloid leukemia. Int J Hematol [Internet]. 2012 Jul [citado 2013 Oct 25];96(1):[about 9 p.]. Available from: http http://www.ncbi.nlm.nih.gov/pubmed/22674382
Nishioka C, Ikezoe T, Yang J, Udaka K, Yokoyama A causes epigenetic alterations of PTEN gene via upregulation of DNA methyltransferases and polycomb group proteins. Blood Cancer J. 2011 Dec;1(12):e48.
Enlaces refback
- No hay ningún enlace refback.










 La Revista está: Certificada por el CITMA
La Revista está: Certificada por el CITMA Acreditados como: "Web de Interés Sanitario"
Acreditados como: "Web de Interés Sanitario"
